虽然是课上第一次学(噢不对,应该是在素红奶奶课上第一次学),但实际上之前已经学过很多次,现在对其数学原理进行进一步分析:
聚类分析属于非监督分类,也就是说基本上无先验知识可依据或参考。 聚类分析根据模式之间的相似性对模式进行分类,对一批没有标出类别的模式样本集,将相似的归为一类,不相似的归为另一类。
相似性
对于特征向量$\mathbf{X} = [x_{1},x_{2}, \dots x_{n}]^T$,将特征空间中向量与向量间的距离作为模式相似性的一种测量方法。以“距离”作为模式分类的一种依据(考)。除此以外还有基于密度的测量等方法。
教材主要介绍的是基于距离的聚类,故以下内容均在此前提下展开
聚类分析也会与数据的分布有关,如果数据是成多簇分布的,那么容易用距离函数进行分类;如果数据均在同一簇,则难以聚类分析
相似性测度(距离测量)
欧氏距离
\[D(\mathbf{X_{i}},\mathbf{X_{j}})= \vert \vert \mathbf{X_{i}} - \mathbf{X_{j}} \vert\vert = \sqrt{ (\mathbf{X_{i}-\mathbf{X_{j}}})^T(\mathbf{X_{i}-\mathbf{X_{j}}}) }\]- 注意由于存在量纲的影响,需要对数据进行标准化,可以统一成标准正态分布或以下形式:
马氏距离
|  |
常用平方形式表示,设$\mathbf{X}$为模式向量,$\mathbf{M}$为某类模式的均值向量,$\mathbf{C}$为该模式总体的协方差矩阵,则马氏距离定义为 \(D^2 = (\mathbf{X}-\mathbf{M})^TC^{-1}(\mathbf{X}-\mathbf{M})\)
其中,$\mathbf{C}$的计算方式为: \(\begin{aligned} \mathbf{C}=&E\{(\mathbf{X-M})(\mathbf{X-M})^T\}=E\begin{bmatrix} (x_{1}-m_{1})\\(x_{2}-m_{2})\\ \vdots \\ (x_{n}-m_{n}) \end{bmatrix}[(x_{1}-m_{1})\quad (x_{2} - m_{2}) \cdots (x_{n}-m_{n})] \\ = &\begin{bmatrix} &E(x_{1}-m_{1}) (x_{1}-m_{1}) &\cdots &E(x_{1}-m_{1}) (x_{n}-m_{n}) \\ &\vdots &\ddots &\vdots \\ &E(x_{n}-m_{n}) (x_{1}-m_{1}) &\cdots &E(x_{n}-m_{n}) (x_{n}-m_{n}) \end{bmatrix} \\ = &\begin{bmatrix} &\sigma_{11}^2 &\cdots &\sigma_{1n}^2\\ &\vdots &\ddots &\vdots \\ &\sigma_{n1}^2 &\cdots &\sigma_{nn}^2 \end{bmatrix} \end{aligned}\)
马氏距离的优点是排除了模式样本之间的相关性影响(考)。 例如我们取一个模式特征向量,可能其中有九个分量反映的是同一特征$A$,而只有一个分量反映特征$B$,这时如用欧氏距离计算,则主要反映了特征$A$,而用马氏距离计算则可避免这个缺点。 当$\mathbf{C}$为单位矩阵$\mathbf{I}$时,马氏距离等同于欧氏距离。
明氏距离
\(D_{m}(\mathbf{X_{i},\mathbf{X_{j}}})=\left[ \sum_{k=1}^{n}\vert x_{ik}-x_{jk}\vert^m \right]^{1/m}\) 当$m=2$时,明式距离即为欧式距离 当$m=1$时,有 \(D_{m}(\mathbf{X_{i},\mathbf{X_{j}}})= \sum_{k=1}^{n}\vert x_{ik}-x_{jk}\vert\) 此时即为曼哈顿距离
汉明距离
如果模式向量各分量仅取1或(-1),即为二值模式。 用汉明距离来衡量相似性 \(D_{h}(\mathbf{X_{i}},\mathbf{X_{j}})=\frac{1}{2}\left( n-\sum_{k=1}^{n}x_{ik} \cdot x_{jk} \right)\) 如果各分量取值均不同,则汉明距离为$n$;若各分量取值均相同,则汉明距离为0
角度(余弦)相似度
\[\cos \theta = S(\mathbf{X_{i},X_{j}}) = \frac{\mathbf{X_{i}^T X_{j}}}{\vert \vert \mathbf{X_{i}} \vert\vert \cdot \vert\vert\mathbf{X_{j}}\vert\vert}\]- 越接近1代表相似度越大
- 模式向量$\mathbf{X_{i},X_{j}}$之间的夹角余弦,也是对应的两个单位向量的点积
- 对坐标系旋转、放大缩小不变
- 当取值仅为01二值时,$\mathbf{X_{i}^T X_{j}}$的值表示两向量共有的特征数目,而$\vert \vert \mathbf{X_{i}}\vert\vert \cdot \vert\vert\mathbf{X_{j}}\vert\vert = \sqrt{ (\mathbf{X_{i}^T X_{i}})(\mathbf{X_{j}^T X_{j}}) }$表示两向量中具有特征数目的几何平均。于是$S(\mathbf{X_{i},X_{j}})$表示两向量中具有共有特征数目的相似性测度
Tanimoto相似度(考)
Tanimoto测度(通常称为Tanimoto系数或Tanimoto相似度)是用于衡量两个样本(通常是向量或集合)相似性的一种指标。它是Jaccard相似系数的一种推广,尤其常用于二值向量、集合、或化学分子结构的比较。在特定领域,如化学信息学,它被广泛用于比较分子指纹特征的相似性。
定义:
给定两个集合 $A$ 和 $B$,Tanimoto系数的定义与Jaccard相似系数类似,公式为:
\[T(A, B) = \frac{|A \cap B|}{|A \cup B|}\]其中:
-
$ A \cap B $ 是集合 $A$ 和 $B$ 的交集的大小,表示它们的共同元素的数量。 -
$ A \cup B $ 是集合 $A$ 和 $B$ 的并集的大小,表示它们的所有不同元素的总数。
对于二进制向量(0和1构成的向量),Tanimoto系数可以推广为如下公式:
\[T(\mathbf{x}, \mathbf{y}) = \frac{\mathbf{x} \cdot \mathbf{y}}{|\mathbf{x}|^2 + |\mathbf{y}|^2 - \mathbf{x} \cdot \mathbf{y}}=\frac{共有的特征数}{占有的特征数目的总数}\]其中:
- $\mathbf{x} \cdot \mathbf{y}$ 表示两个向量的点积。
-
$ \mathbf{x} ^2$ 和 $ \mathbf{y} ^2$ 是向量 $\mathbf{x}$ 和 $\mathbf{y}$ 的范数(向量长度)的平方。
解释:
- 值范围:Tanimoto系数的值在0和1之间:
- 当两个集合(或向量)完全相同,Tanimoto系数为1。
- 当两个集合没有任何共同元素,Tanimoto系数为0。
- 应用:Tanimoto系数经常用于以下领域:
- 集合相似性:用于评估两个集合的相似度,尤其是在文档分类或推荐系统中。
- 化学信息学:用于比较分子指纹,帮助寻找具有相似化学性质的分子。
- 机器学习和数据挖掘:在特征选择和相似性度量中,尤其是在稀疏向量或二值数据(如推荐系统的用户行为数据)中使用。
聚类准则
根据相似性测度确定的,衡量模式之间是否相似的标准。即把不同模式聚为一类,还是归为不同类的准则(考)
确定方式:
- 阈值准则:根据规定的距离阈值进行分类
- 函数准则:根据聚类准则函数进行分类的准则
函数准则
误差平方和 \(J=\sum_{j=1}^{c}\sum_{\mathbf{X}\in S_{j}} \vert\vert\mathbf{X-M_{j}}\vert\vert^2\) 其中,$c$表示共有$c$个模式类,$\mathbf{M_{j}}=\frac{1}{N}\sum_{\mathbf{X}\in S_{j}}\mathbf{X}$,为$S_{j}$中样本的均值向量,$N_{j}$为$\mathbf{S_{j}}$中的样本数目
当$J$达到极小时,说明达到了满意的分类效果。这种准则通常称为最小方差划分 适用于各类样本密集且数量相差不多,而不同类间样本又明显分开的情况
基于距离阈值的聚类算法
近邻聚类法
- 任取样本$X_{i}$ 作为第一个聚类中心的初始值,如令$Z_{1}=X_{1}$ 。
- 计算样本$X_{2}$ 到$Z_{1}$ 的欧氏距离$D_{21}=\vert\vert X_{2}-Z_{1}\vert\vert$, 若$D_{21}>T$,定义一新的聚类中心$Z_{2} = X_{2}$ ; 否则 $X_{2} ∈$以$Z_{1}$为中心的聚类。
- 假设已有聚类中心$Z_{1},Z_{2}$,计算$D_{31}=\vert\vert X_{3}-Z_{1}\vert\vert$和$D_{32}=\vert\vert X_{3}-Z_{2}\vert\vert$ 若$D_{31}>T$且$D_{32}>T$,则建立第三个聚类中心$Z_{3}=X_{3}$ 否则 $X_{3} ∈$离$Z_{1}$和$Z_{2}$最近的聚类。
算法特点
- 局限性:很大程度上依赖于第一个聚类中心的位置选择、待分类模式样本的排列次序、距离阈值T的大小以及样本分布的几何性质等。
- 优点:计算简单。(一种虽粗糙但快速的方法)
最大最小距离算法(小中取大)
- 选任意一模式样本做为第一聚类中心$Z_{1}$
- 选择离$Z_{1}$距离最远的样本作为第二聚类中心$Z_{2}$
- 逐个计算各模式样本与已确定的所有聚类中心之间的距离,并选出其中的最小距离。例当聚类中心数k=2时,计算$D_{i_{1}}=\vert\vert x_{1}-z_{1}\vert\vert, D_{i_{2}}= \vert\vert x_{1} - z_{2} \vert\vert$,找到$D_{i_{1}}, D_{i_{1}}$最小值 $\min (D_{i_{1}}, D_{i_{2}})$
- 在所有最小距离中选出最大距离,如该最大值达到$\vert\vert Z_{1}-Z_{2}\vert\vert$的一定分数比值(阈值$T$) 以上,则相应的样本点取为新的聚类中心,返回3;否则,寻找聚类中心的工作结束。 例如$k=2$,若$\max{\min(D_{i_{1}},D_{i_{2}})}>\theta\vert\vert Z_{1}-Z_{2}\vert\vert$,则$Z_{3}$存在
- 重复步骤3,4,直到没有新的聚类中心出现为止。
- 将样本${X_{i}, i=1,2,\dots,N}$按最近距离划分到相应聚类中心对应的类别中。

层次聚类法
每个样本先自成一类,然后按距离准则逐步合并,减少类数。
算法描述
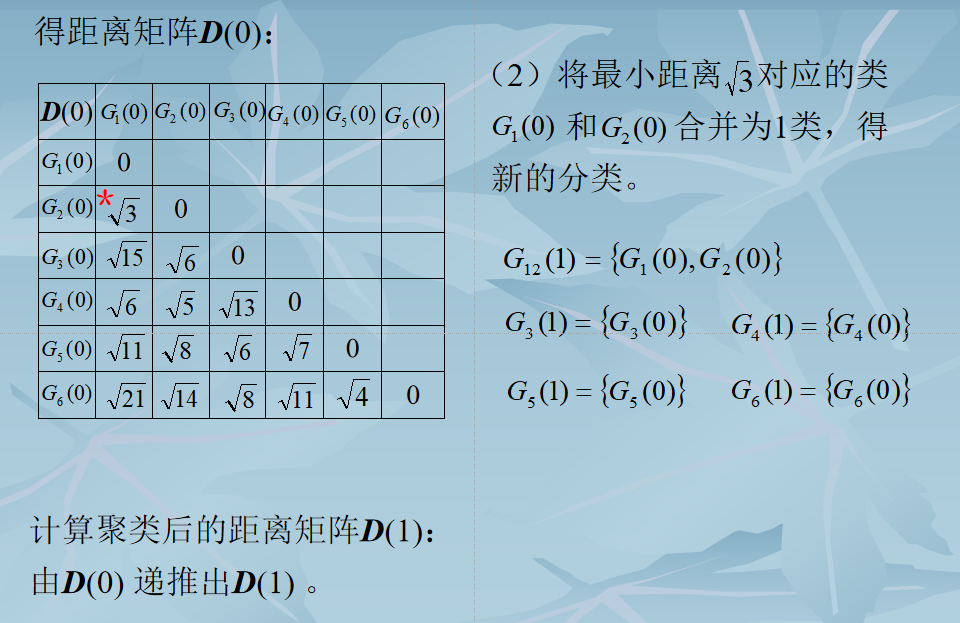
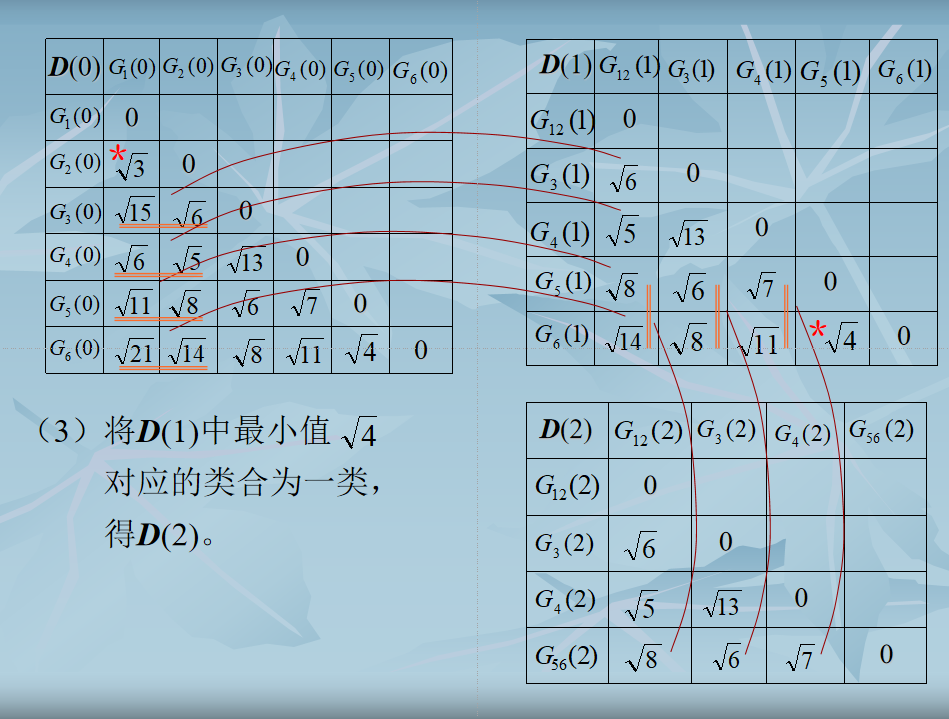
- N个初始模式样本自成一类,即建立$N$类: \(G_{1}(0),G_{2}(0),\dots,G_{N}(0)\) 计算各类之间(即各样本间)的距离,得一$N×N$维距离矩阵D(0)。“0”表示初始状态。
- 假设已求得距离矩阵$D(n)$(n为逐次聚类合并的次数),找出$D(n)$中的最小元素,将其对应的两类合并为一类。由此建立新的分类:$G_{1}(n+1),G_{2}(n+1)$
- 计算合并后新类别之间的距离,得$D(n+1)$。
- 跳至第2步,重复计算及合并。
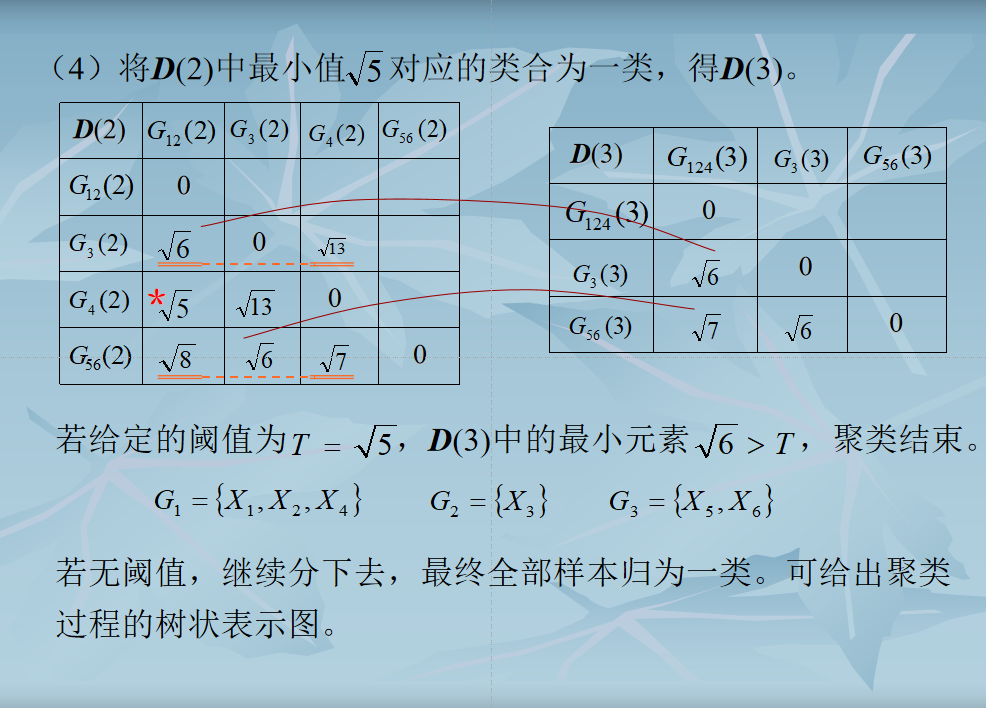
结束条件
- 取距离阈值$T$,当$D(n)$的最小分量超过给定值 $T$ 时,算法停止。所得即为聚类结果。
- 或不设阈值T,一直将全部样本聚成一类为止,输出聚类的分级树
类间距离计算
最短距离法
\(D_{HK}=\min\{D(X_{H},X_{k})\} X_{H} \in H, X_{K} \in K\) $D(X_{H},X_{K})$ H类中的某个样本$X_{H}$和K类中的某个样本$X_{K}$之间的欧式距离
最长距离法
\[D_{HK}=\max\{D(X_{H},X_{k})\} X_{H} \in H, X_{K} \in K\]中间距离法
如果K类由I类和J类合并而成,则H和K类之间的距离为
\[D_{HK}=\sqrt{ \frac{1}{2}D_{HI}^2 + \frac{1}{2} D_{HJ}^2 - \frac{1}{4} D_{IJ}^2}\]重心法
将每类中包含的样本数考虑进去。若$I$类中有$n_{I}$个样本,$J$类中有$n_{J}$个样本,则类与类之间的距离递推式为
\(D_{HK}=\sqrt{ \frac{n_{I}}{n_{I}+n_{J}}D_{HI}^2 + \frac{n_{J}}{n_{I}+n_{J}} D_{HJ}^2 - \frac{n_{I}n_{J}}{(n_{I}+n_{J})^2} D_{IJ}^2}\)
类平均距离
\(D_{HK}=\sqrt{ \frac{1}{n_{H}n_{K}}\sum_{i \in H, j \in K} d_{ij}^2 }\) $d_{ij}^2$: H类任一样本$X_{i}$和K类任一样本$X_{j}$之间的欧氏距离平方。
(考)

Ward’s 簇间距离
\[\begin{equation} \begin{aligned} d(C_{k},C_{j}) &= \sqrt{ 2\times \left( \sum_{x \in C_{k} \cup C_{j}} dist(x, \mu_{C_{k} \cup C_{j}})^2 - \left( \sum_{x \in C_{k}}dist(x, \mu_{C_{k}})^2+ \sum_{x \in C_{j}}dist(x, \mu_{C_{j}})^2\right) \right)} \\ &=\sqrt{ \frac{2 \cdot count(C_{k}) \cdot count(C_{j})}{count(C_{k}) + count(C_{j})} }\cdot dist(\mu_{C_{k}},\mu C_{j}) \\&= \sqrt{ 2\times (SST(C_{k}\cup C_{j})-(SST(C_{k})+SST(C_{j}))) } \end{aligned} \end{equation}\]clustering_algorithms = (
('Single linkage', 'single'),
('Average linkage', 'average'),
('Complete linkage', 'complete'),
('Ward linkage', 'ward'),
)
for name, method in clustering_algorithms:
# 绘制树形图
fig, ax = plt.subplots()
plt.title(name)
dend = dendrogram(linkage(X,
method = method))
# 层次聚类
cluster = AgglomerativeClustering(n_clusters=3,
metric='euclidean',
linkage=method)
# 完成聚类预测
Z = cluster.fit_predict(X)
# 可视化聚类结果
fig, ax = plt.subplots()
plt.title(name)
# 可视化散点图
plt.scatter(x=X[:, 0], y=X[:, 1], c=Z, alpha=1.0,
linewidth = 1, edgecolor=[1,1,1])
ax.set_xticks(np.arange(4, 8.5, 0.5))
ax.set_yticks(np.arange(1.5, 5, 0.5))
ax.set_xlim(4, 8)
ax.set_ylim(1.5, 4.5)
plt.xlabel(iris.feature_names[0])
plt.ylabel(iris.feature_names[1])
ax.grid(linestyle='--', linewidth=0.25, color=[0.5,0.5,0.5])
ax.set_aspect('equal')
plt.show()
基于密度的聚类
[!note] 本部分主要介绍DBSCAN,其余方法作为补充
基本概念
DBSCAN算法包含以下几个基本概念:
- $\varepsilon$邻域:对于一个给定的数据点,$\varepsilon$邻域是指在其半径$\varepsilon$范围内的所有数据点的集合。$\varepsilon$是一个用户定义的距离参数。
- MinPts阈值(min_samples):MinPts是定义一个点是否为核心点的阈值。一个点的$\varepsilon$邻域内至少包含MinPts个数据点,该点才被认为是核心点。
- 核心点:如果一个点的$\varepsilon$邻域内包含至少MinPts个点,则该点是一个核心点。
- 边界点:边界点是指在核心点的$\varepsilon$邻域内,但自身的$\varepsilon$邻域内的点数小于MinPts的点。
- 噪声点:既不是核心点也不是边界点的点被称为噪声点。
- 密度可达:如果点P在点Q的$\varepsilon$邻域内,并且Q是核心点,那么点P是从点Q密度可达的。
- 密度连接:如果存在一个点链,使得每一对相邻点之间都是密度可达的,那么两个点之间就是密度连接的。
聚类过程
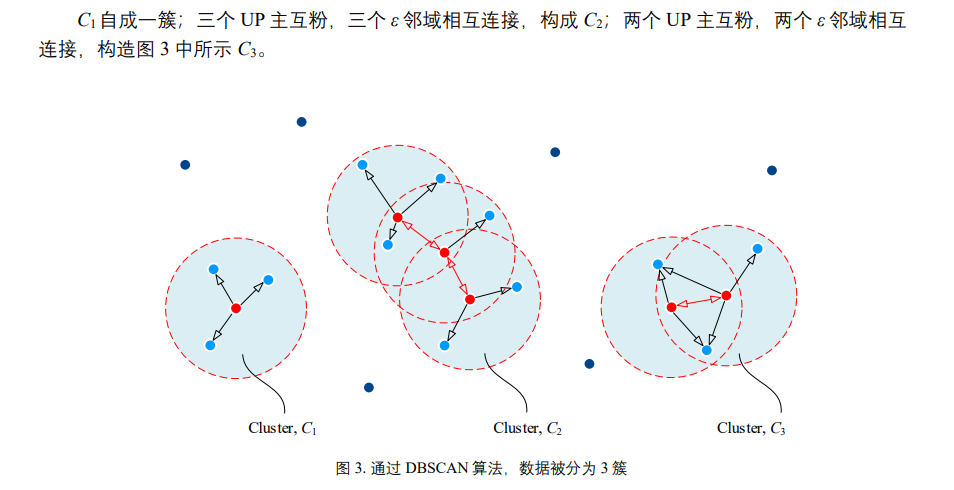
给出平面内8个样本数据点,以每个数据点为中心,$ε$ 为半径扫描整个平面,且定义 min_samples = 4。 发现只有样本点 $x (5)$的 $ε$ 邻域内有 4 个样本点 (包括 $x (5)$自身);因此,$x5$为核心点,$x (2)、x (4)和 x (7)$为 边界点,剩余其他数据点为噪点。
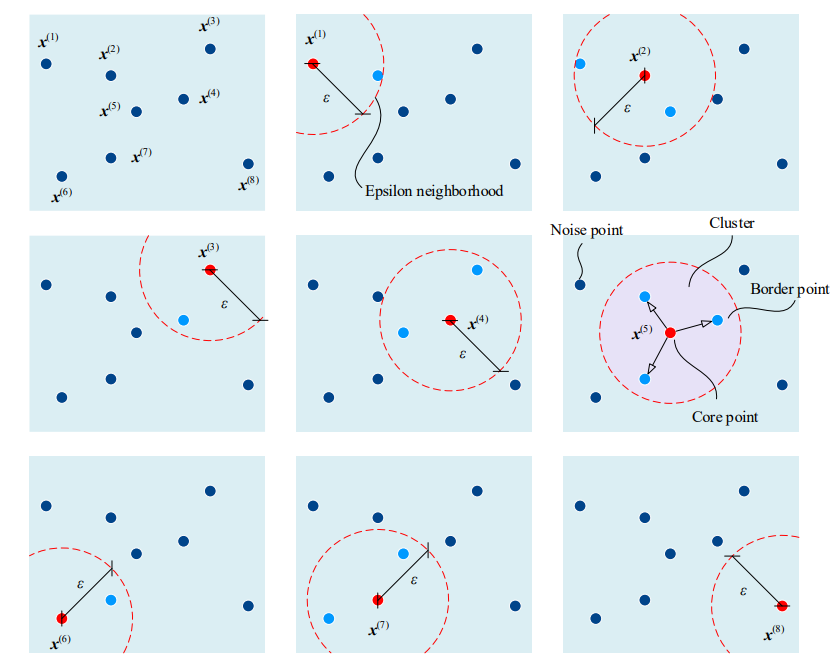
如果一个点既是边界点又是核心点,那么此时会有两个簇连接在一起,以此类推:
调节参数
邻域范围
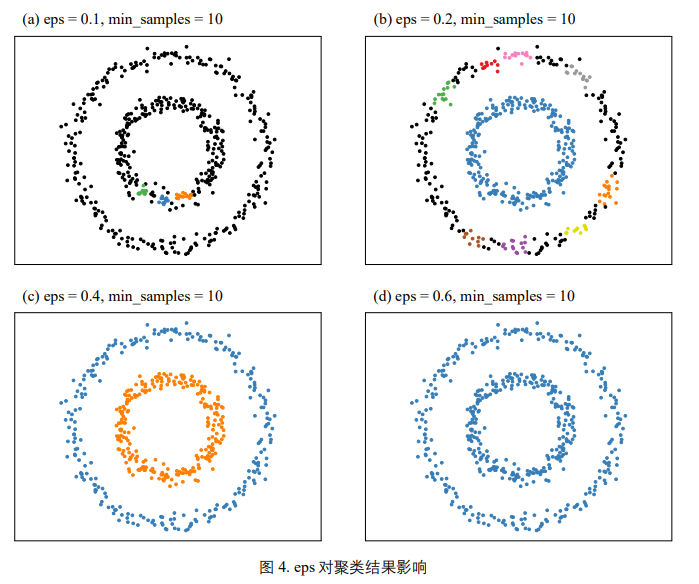
eps 控制邻域范围大小。eps 值选取过大,会导致整个数据集被分为一簇;但是 eps 取值过小,会 导致簇过多且分散,并且标记过多噪音点。
for eps in np.array([0.1,0.2,0.4,0.6]):
dbscan = cluster.DBSCAN(eps=eps,min_samples=10)
y_pred = dbscan.fit_predict(X)
fig, ax = plt.subplots()
colors = np.array(list(islice(cycle(['#377eb8', '#ff7f00', '#4daf4a',
'#f781bf', '#a65628', '#984ea3',
'#999999', '#e41a1c', '#dede00']),
int(max(y_pred) + 1))))
# 增加黑色
colors = np.append(colors, ["#000000"])
# 绘制散点图
plt.scatter(X[:, 0], X[:, 1], s=10, color=colors[y_pred])
plt.title('eps = %0.2f' % eps)
plt.xlim(-2.5, 2.5)
plt.ylim(-2.5, 2.5)
plt.xticks(())
plt.yticks(())
plt.axis('equal')
plt.show()
邻域内样本个数
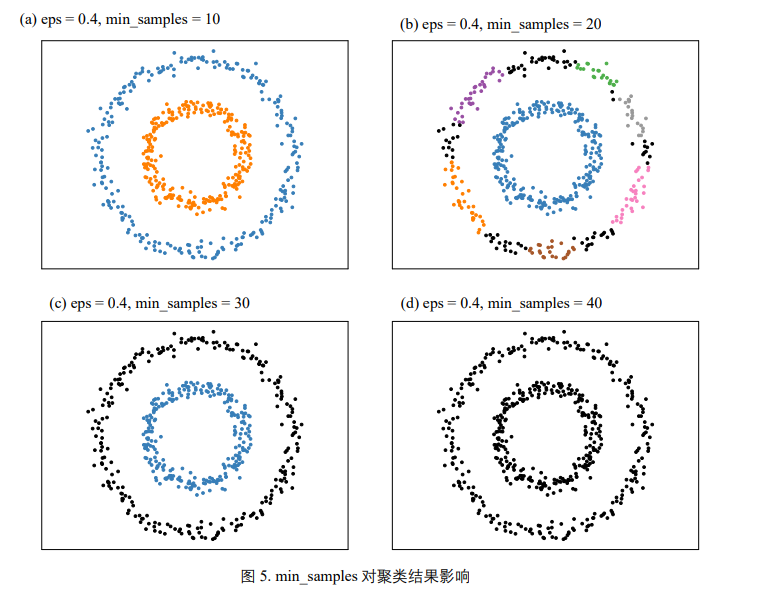
min_samples 调节 DBSCAN 算法对噪声的容忍度;当数据噪音过大时,应该适当提高 min_samples。 k 均值和 GMM 聚类算法需要预先声明聚类数量;但是,DBSCAN 则不需要。DBSCAN 聚类不需要 预设分布类型,不受数据分布影响,且可以分辨离群数据。 DBSCAN 算法对 eps 和 min_samples 这两个初始参数都很敏感;协同调节 eps 和 min_samples 两个参数显得非常重要。
优点:
- 无需指定簇的个数:与K-means等算法不同,DBSCAN不需要预先指定聚类的数量。
- 处理噪声:能够有效识别并处理噪声点。
- 发现任意形状的簇:能够发现任意形状的簇,而不仅仅是圆形或球形的簇。
缺点:
- 参数敏感:算法对参数Eps和MinPts较为敏感,选择不当会影响聚类结果。
- 性能问题:在高维数据集上性能不佳,计算Eps邻域的时间复杂度为O(n^2),对于大规模数据集不够高效。
- 不适用于不同密度的簇:如果数据集中的簇有显著不同的密度,DBSCAN可能无法很好地识别所有簇。
- 对密度分布较为均匀的数据集,可能会出现聚类失效
动态聚类
K-means(考计算)
比如,二聚类问题有两个簇质心$\mu_{1}$和$\mu_{2}$ 如果以欧式距离进行距离度量,那么离质心$\mu_{1}$更近的点,被划分为$C_{1}$簇;反之被划分为$C_{2}$簇

由于采用欧氏距离,图 1中簇质心 $µ_{1}$ 和 $µ_{2}$等高线为两组同心圆;同心圆颜色相同,代表距离簇质心 $µ_{1}$ 和 $µ_{2}$距离相同。因此,同色同心圆的交点位于决策边界上。
实际上就是质心之间的中垂线 三聚类时更加明显:

优化目标
将所有样本点划分为$K$簇,并使得簇内距离平方和最小 \(\arg \min_{c} \sum_{k=1}^{K} \sum_{x \in C_{k}} ||x - \mu_{k}||^2\)
对于每一个聚类集,将准则函数定义为 \(J_{j} = \sum_{x \in C_{j}} ||x - \mu_{j}||^2\) 由于要使得准则函数最小,对其求偏导 \(\frac{\partial}{\partial \mu_{j}} \sum_{x \in C_{j}} ||x - \mu_{j}||^2 = \frac{\partial}{\partial \mu_{j}} \sum_{x \in C_{j}}(x-\mu_{j})^T(x-\mu_{j})=0\) 需要对其进行展开 \(\frac{\partial}{\partial \mu_{j}} \sum_{x \in C_{j}}(x-\mu_{j})^T(x-\mu_{j})=\frac{\partial}{\partial \mu_{j}} \sum_{x \in C_{j}}(x^Tx-2x^T\mu_{j}+\mu_{j}^2)=\sum_{x \in C_{j}}(-2x^T+2\mu_{j})=0\) 其中,$-2$可以消去,再次进行展开 \(\sum_{x \in C_{j}} x^T = \sum_{x \in C_{j}} \mu_{j}\) 由于$\mu_{j}$与$x$无关,故可提到求和之外,右侧可化简 \(\sum_{x \in C_{j}} x^T = |C_{j}| \mu_{j}\) 解得 \(\mu_{j} = \frac{1}{|C_{j}|}\sum_{x \in C_{j}} x^T\) 说明$C_{j}$类的聚类中心应为该类样本的均值
迭代过程
此处$Z_i$和$\mu_{i}$意思等同,只是因为老师强制要求符号这么写,所以进行记录
- 任选K个初始聚类中心:$Z_{1}(1)$, $Z_{2}(1)$,…, $Z_{K}(1)$。(括号内序号表示迭代运算的次序号)
- 按最小距离原则将其余样品分配到K个聚类中心中的某一个,即:
- 计算各个聚类中心的新向量值:$Z_{j}(k+1),j=1,2,\dots,K$
- 如果$Z_{j}(k+1) \neq Z_{j}(k), j=1,2,\dots,K$,回到(2),重新分类迭代计算;如果取等,此时算法收敛





代码1 (比较复杂可以不看)
# 导入鸢尾花数据
iris = datasets.load_iris()
# 取出鸢尾花前两个特征
X_train = iris.data[:, :2]
y_train = iris.target
# 创建KMeans对象
kmeans = KMeans(n_clusters=3, n_init = 'auto')
# 使用KMeans算法训练数据
kmeans.fit(X_train)
# 生成网格数据
plot_step = 0.02
xx, yy = np.meshgrid(np.linspace(4, 8, int(4/plot_step + 1)),
np.linspace(1.5, 4.5, int(3/plot_step + 1)))
# 使用KMeans模型对网格中的点进行预测
# 并将预测结果整形成与网格相同形状的矩阵
Z = kmeans.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
fig, ax = plt.subplots()
# plot regions
plt.contourf(xx, yy, Z, cmap=cmap_light)
# plot sample data
plt.scatter(x=X_train[:, 0], y=X_train[:, 1], color=np.array([0, 68, 138])/255., alpha=1.0, linewidth = 1, edgecolor=[1,1,1])
# plot decision boundaries
plt.contour(xx, yy, Z, levels=[0,1,2], colors=np.array([0, 68, 138])/255.)
# plot centroids
centroids = kmeans.cluster_centers_
plt.scatter(centroids[:, 0], centroids[:, 1], marker="x", s=100, linewidths=1.5, color="k")
ax.set_xticks(np.arange(4, 8.5, 0.5))
ax.set_yticks(np.arange(1.5, 5, 0.5))
ax.set_xlim(4, 8)
ax.set_ylim(1.5, 4.5)
plt.xlabel(iris.feature_names[0])
plt.ylabel(iris.feature_names[1])
ax.grid(linestyle='--', linewidth=0.25, color=[0.5,0.5,0.5])
ax.set_aspect('equal')
plt.show()
1. np.meshgrid() 和生成网格数据
np.meshgrid 是用来生成二维坐标网格的函数,它会根据提供的输入向量生成两个矩阵,表示网格点的横坐标和纵坐标。
解释代码:
plot_step = 0.02
xx, yy = np.meshgrid(np.linspace(4, 8, int(4/plot_step + 1)),
np.linspace(1.5, 4.5, int(3/plot_step + 1)))
plot_step = 0.02:表示网格的间距,每个网格点之间相差0.02。np.linspace(4, 8, int(4/plot_step + 1)):这部分生成从 4 到 8 之间的等间隔点,步长为plot_step。生成的点的个数是int(4/plot_step + 1),即从 4 到 8 之间一共生成(8 - 4) / 0.02 + 1 = 201个点。这个向量表示网格在 x 轴上的坐标。np.linspace(1.5, 4.5, int(3/plot_step + 1)):同理,生成从 1.5 到 4.5 之间的等间隔点,表示 y 轴上的坐标。np.meshgrid():它会将两个向量(x 和 y 坐标)组合成一个二维网格,这样可以方便地进行二维平面上的计算。xx表示网格中每个点的 x 坐标,yy表示网格中每个点的 y 坐标。
例如,xx 和 yy 的形状将会是相同的,它们都是 $201 \times 151$ 的矩阵。xx 的每一行都表示 x 坐标的值,而 yy 的每一列都表示 y 坐标的值。
2. np.c_[xx.ravel(), yy.ravel()]
np.c_ 是 NumPy 用于水平拼接数组的功能,它将多个数组按列进行拼接。
解释代码:
Z = kmeans.predict(np.c_[xx.ravel(), yy.ravel()])
xx.ravel():将矩阵xx拉平成一维数组,按照行优先顺序展平。yy.ravel():同样,将yy展平成一维数组。xx.ravel()和yy.ravel()现在是两个一维数组,它们的长度均为 (201 \times 151 = 30351)。np.c_[xx.ravel(), yy.ravel()]:将xx.ravel()和yy.ravel()按列拼接在一起,形成一个形状为 (30351 \times 2) 的二维数组。每一行代表网格中的一个点,第一列是 x 坐标,第二列是 y 坐标。 通过np.c_[xx.ravel(), yy.ravel()],我们创建了一个包含所有网格点坐标的二维数组,便于后续使用 KMeans 模型进行预测。
总结:
np.meshgrid()生成了二维平面的网格坐标。np.c_[xx.ravel(), yy.ravel()]将网格中的每一个点的 x 和 y 坐标组合成一个 $30351 \times 2$ 的数组,表示网格中所有点的坐标,供kmeans.predict进行分类。
代码2 常用
# pandas导入
df = pd.read_csv('./ch1ex1.csv')
points = df.values
xs = points[:, 0]
ys = points[:, 1]
model = KMeans(n_clusters = 3, n_init = 'auto')
model.fit(points)
labels = model.predict(points)
print(labels) # 打印每个点被分到的类
# 聚类中心
centroids = model.cluster_centers_
centroids_x = centroids[:, 0]
centroids_y = centroids[:, 1]
# 原始数据点
xs = points[:, 0]
ys = points[:, 1]
# 建立装饰和颜色
mk0 = ['o', ',', 'v']
cs0 = ['r', 'g', 'b']
mk1 = []
cs1 = []
for e in labels:
mk1.append(mk0[e])
cs1.append(cs0[e])
# 画点和质心
plt.figure(figsize=(10, 6), dpi = 120)
plt.subplot(111)
for x, y, cr, m ,in zip(xs, ys, cs1, mk1):
plt.scatter(x, y, edgecolors=cr, facecolors = 'none', marker=m)
plt.scatter(centroids_x, centroids_y, marker = 'X', s = 200, c = 'k')
plt.show()
肘部系数
用于判断合适的聚类簇值K \(SSE(X|K)=\sum_{k=1}^{K}SSE(C_{k})=\sum_{k=1}^{K}\sum_{x \in C_{k}}||x-\mu_{k}||^2\) 曲线的拐点对应着接近最优的K值(SSE减小量、计算量以及分类效果的权衡)。
seeds_df = pd.read_csv('./seeds-less-rows.csv')
# print(seeds_df.grain_variety.value_counts())
varieties = list(seeds_df['grain_variety'])
samples = seeds_df.values
#print(len(samples))
ks = range(1, 6)
inertias = []
for k in ks:
#Create a KMeans instance with k clusters: model
model = KMeans(n_clusters=k, n_init = 'auto')
#Fit model to samples
model.fit(samples)
# Append the inertia to the list of inertias
inertias.append(model.inertia_)
plt.figure(figsize=(10, 6), dpi=80)
plt.subplot(111)
#plot ks vs inertias
plt.plot(ks, inertias, '-o')
plt.xlabel('number of clusters, k')
plt.ylabel('inertia')
plt.xticks(ks)
plt.show()

轮廓图:选定聚类簇值
轮廓图上每一条线代表的是轮廓系数: \(s_{i}=\frac{b_{i}-a_{i}}{\max \{a_{i},b_{i}\}}\) 其中,$a_{i}$为簇内不相似度,$b_{i}$为簇间不相似度
簇内不相似度
样本$i \in C_{k}$到同簇其他样本$j(j \in C_{k}, i \neq j)$距离的平均值: \(a_{i}=\frac{1}{count(C_{k})-1}\sum_{j \in C_{k},i\neq j} {d_{i,j}}\) $d_{i,j}$为样本$i$和$j$之间的距离,$a_{i}$越小1,说明越应该被划分到$C_{k}$簇
簇间不相似度
样本$i \in C_{k}$到其他簇样本$j(j \in C_{m}, C_{m} \neq C_{k})$距离的平均值: \(b_{i} = \min \frac{1}{count(C_{m})}\sum_{j \in C_{m}}{d_{i,j}}\)
[!note] 当簇数超过2时,$b_{i}$需要在不同簇中找到最小值
计算轮廓系数的函数为 sklearn.metrics.silhouette_score
yellowbrick.cluster.SilhouetteVisualizer 函数绘制轮廓图
from sklearn.cluster import KMeans
from yellowbrick.cluster import SilhouetteVisualizer
from sklearn.metrics import silhouette_score
kmeans = KMeans(n_clusters=n_clusters, random_state=10)
cluster_labels = kmeans.fit_predict(X)
silhouette_avg = silhouette_score(X, cluster_labels)
print("For n_clusters =", n_clusters, "The average silhouette_score is :", silhouette_avg)
# For n_clusters = 3 The average silhouette_score is : 0.445052569
visualizer = SilhouetteVisualizer(kmeans, colors='yellowbrick')
visualizer.fit(X)
# Fit the data to the visualizer
visualizer.show()
沃罗诺伊图
质心中垂线相交分割区域

迭代自组织(ISODATA)
与K-均值算法的比较
- K-均值算法通常适合于分类数目已知的聚类,而ISODATA算法则更加灵活;
- 从算法角度看, ISODATA算法与K-均值算法相似,聚类中心都是通过样本均值的迭代运算来决定的;
- ISODATA算法加入了一些试探步骤,并且可以结合成人机交互的结构,使其能利用中间结果所取得的经验更好地进行分类。
- K-均值算法的聚类中心个数不变;ISODATA的聚类中心个数变化。

算法步骤
第一步:输入$N$个模式样本${x_{i},i=1,2,…,N}$
预选$N_{c}$个初始聚类中心${z_{1},z_{2},…z_{N_{c}}}$,它可以不等于所要求的聚类中心的数目,其初始位置可以从样本中任意选取。
预选: $K$ = 预期的聚类中心数目; $θ_{N}$ = 每一聚类域中最少的样本数目,若少于此数即不作为一个独立的聚类; $θ_{S}$ = 一个聚类域中样本距离分布的标准差;标准差向量的每一分量反映样本在特征空间的相应维上,与聚类中心的位置偏差(分散程度)。要求每一聚类内,其所有分量中的最大分量应小于$θ_{S}$,否则该类将被分裂为两类。 $θ_{c}$ = 两个聚类中心间的最小距离,若小于此数,两个聚类需进行合并; $L$ = 在一次迭代运算中可以合并的聚类中心的最多对数; $I$ = 迭代运算的次数。
| 第二步:将$N$个模式样本分给最近的聚类$S_{j}$,假若$D_{j}= | x-z_{j} | =min{‖x−z_{i}‖,i=1,2,⋯N_{c}}$,即$ | x−z_{j} | $的距离最小,则$x∈S_{j}$。 |
第三步:如果$S_{j}$中的样本数目$S_{j}<θ_{N}$,则取消该样本子集,此时$N_{c}$减去1。 (以上各步对应基本步骤(1))
第四步:修正各聚类中心 \(z_{j}=\frac{1}{N_{j}}∑_{x∈S_{j}}x,\ j=1,2,⋯,N_{c}\)
第五步:计算各聚类域$S_{j}$中模式样本与各聚类中心间的平均距离 \(\bar{D_{j}}=\frac{1}{N_{j}}∑_{x∈S_{j}}∥x−z_{j}∥,j=1,2,⋯,N_{c}\)
第六步:计算全部模式样本和其对应聚类中心的总平均距离 \(\bar{D}=\frac{1}{N}\sum_{j=1}^{N_{c}}N_{j}\bar{D_{j}}\) (以上各步对应基本步骤(2))
第七步:判别分裂、合并及迭代运算
- 若迭代运算次数已达到$I$次,即最后一次迭代,则置$θ_{c} =0$,转至第十一步。
- 若$N_{c} ≤ \frac{K}{2}$,即聚类中心的数目小于或等于规定值的一半,则转至第八步,对已有聚类进行分裂处理。
- 若迭代运算的次数是偶数次,或$N_c≥2K$,即聚类中心数目大于或等于希望数的两倍,不进行分裂处理,转至第十一步(合并);否则(即既不是偶数次迭代,又不满足$N_{c}≥2K$),转至第八步,进行分裂处理。
(以上对应基本步骤(3))

第八步:计算每个聚类中样本距离的标准差向量$σ_{j}=[σ_{j_{1}},σ_{j_{2}},…,σ_{jn}]T$ 其中向量的各个分量为\(\sigma_{ji}=\sqrt{ \frac{1}{N_{j}}\sum_{x \in S_{j}}(x_{ji}-z_{ji})^2 }=\sqrt{ 方差 }\) 式中,$i = 1, 2, …, n$为样本特征向量的维数,$j = 1, 2, …, N_{c}$为聚类数,$N_{j}$为$S_j$中的样本个数。
第九步:求每一标准差向量${σ_{j}, j = 1, 2, …, N_{c}}$中的最大分量,以${σ_{jmax}, j = 1, 2, …, Nc}$代表。
第十步:在任一最大分量集${σ_{jmax}, j = 1, 2, …, N_{c}}$中,若有$σ_{jmax}>θ_{S}$(标准差阈值),说明$S_{j}$类样本在对应方向上的标准差大于允许的值,同时又满足如下两个条件之一:
- $\bar{D_{j}}>\bar{D}$和$N_{j} > 2(θ_{N} + 1)$,即类内平均距离大于总体平均距离,且$S_{j}$中样本总数超过规定值一倍以上($\theta_{N}$为每个聚类中的最少样本数);
- $N_{c} ≤ \frac{K}{2}$,即聚类数小于或等于希望数目的一半。
则将 $z_{j}$ 分裂为两个新的聚类中心$Z_{j}^+$和$Z_{j}^-$,且$N_{c}$加1。$Z_{j}^+ = σ_{jmax}$+$kσ_{jmax}$,$Z_{j}^-=σ_{jmax}-kσ_{jmax}$,$0<k\leq{1}$,称为分裂系数。
如果本步骤完成了分裂运算,迭代次数+1,则转至第二步,否则继续。 (以上对应基本步骤(4)进行分裂处理)
合并处理: 第十一步:计算全部聚类中心的距离 \(D_{ij}=||z_{i}−z_{j}||,i=1,2,…,N_{c}−1,j=i+1,…,N_{c}\)
第十二步:比较$D_{ij}$与$θ_{c}$(两聚类中心的最小距离)的值,将$D_{ij} <θ_c$ 的值按最小距离次序递增排列,即 \(\{D_j_{1}},D_{i_{2}j_{2}},…,D_{i_{L}j_{L}}\}\) 式中$D_j_{1}}<D_{i_{2}j_{2}}<…<D_{i_{L}j_{L}}$。
第十三步:将距离为$D_{i_{k}j_{k}}$的两个聚类中心$Z_{ik}$和$Z_{jk}$合并,得新的中心为: \(z^∗_{k}= \frac{1}{N_{ik}+N_{jk}} [N_{ik}z_{ik}+N_{jk}z_{jk}],k=1,2,⋯,L\) 式中,被合并的两个聚类中心向量分别以其聚类域内的样本数加权,使$Z^∗_{k}$为真正的平均向量。 (以上对应基本步骤(5)进行合并处理)
第十四步:如果是最后一次迭代运算(即第$I$次),则算法结束;否则,若需要操作者改变输入参数,转至第一步;若输入参数不变,转至第二步。 在本步运算中,迭代运算的次数每次应加1。






